近日,8040威尼斯陈庆山教授团队辛大伟教授课题组在国际知名学术期刊《Plant, Cell & Environment》(中科院一区TOP期刊,五年IF=7.6)在线发表了题为“Single‐Cell RNA‐Sequencing of Soybean Reveals Transcriptional Changes and Antiviral Functions of GmGSTU23 and GmGSTU24 in Response to Soybean Mosaic Virus”的研究论文,构建了大豆花叶病毒(SMV)侵染后的大豆叶片单细胞转录组图谱,并应用于大豆抗病毒相关基因的挖掘。

文章题目
SMV侵染引起的大豆花叶病毒病是大豆生产上的重要病害,在世界各大豆产区普遍发生,可造成大豆叶片花叶、畸形、坏死,植株矮化,种粒斑驳等多种症状,严重影响大豆产量和品质。研究病毒与寄主植物细胞的互作网络,明确病毒侵染后寄主植物细胞基因表达模式的变化,对于解析SMV的致病机制,培育抗SMV大豆新品种具有重要意义。然而,传统的RNA测序技术只能测定样品中所有细胞的平均基因表达水平,无法反映不同类型细胞间基因表达的差异。单细胞测序技术的出现为克服该问题提供了有力工具。
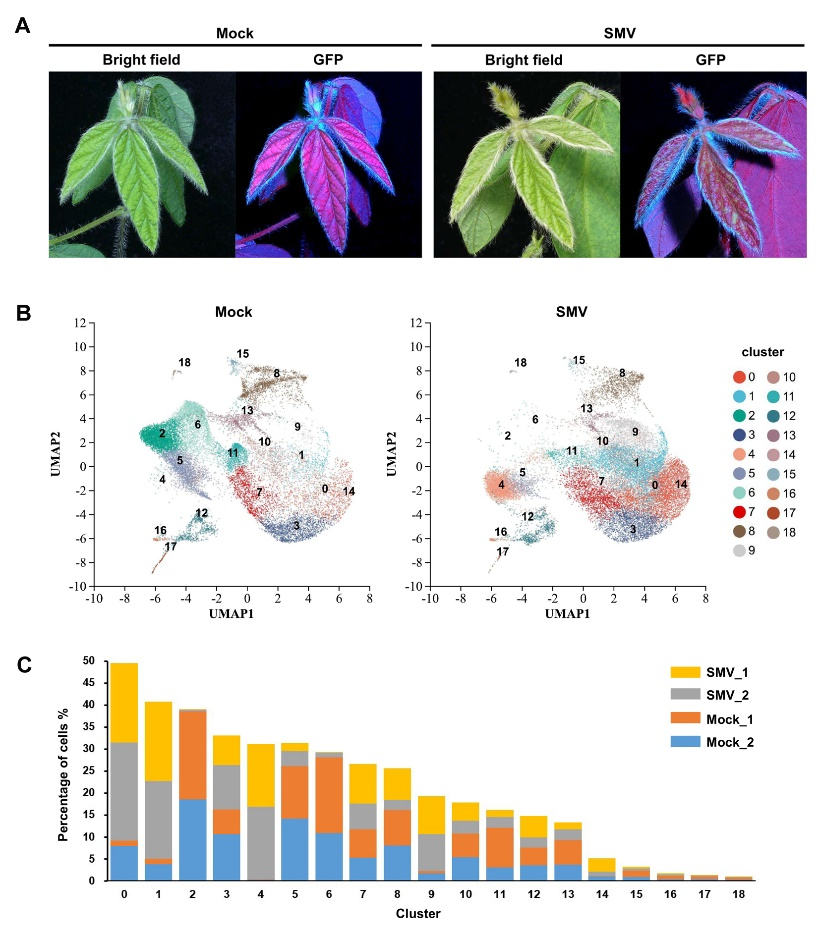
SMV侵染后的大豆叶片单细胞转录组测序
在本研究中,研究人员使用绿色荧光蛋白标记的重组SMV毒源接种感病大豆品种绥农14,在接种后7 d系统叶首次出现绿色荧光时采集系统叶样品进行了单细胞测序。实验共获得50294个细胞的转录本数据,通过降维分析划分出19个cluster,根据已报道的拟南芥Marker基因的大豆同源基因表达情况和RNA原位杂交验证,将其中18个cluster分别注释为表皮、叶肉和维管束3种细胞类型,并证实了Glyma.17g166600、Glyma.15G057600和Glyma.10G039800可以分别作为大豆叶片表皮、叶肉和维管束细胞的新Marker基因。在上述3种细胞中,维管束细胞在SMV侵染后差异表达基因的数量最多,并且显著富集于谷胱甘肽代谢途径。因此,研究人员对大豆谷胱甘肽硫转移酶基因(GmGST)家族进行了进一步分析,共鉴定到103个GmGST基因,其中GmGSTU23和GmGSTU24在上述3种类型细胞中均受SMV侵染诱导而显著上调表达。在本氏烟草中过表达GmGSTU23和GmGSTU24均能显著抑制SMV的增殖。综上所述,本研究为深入解析大豆与SMV的互作机制提供了重要理论依据。
学校植物保护学院宋爽副教授和农学院已毕业硕士王婧为该论文的第一作者,农学院智慧农场技术与系统全国重点实验室辛大伟教授和陈庆山教授为该论文的通讯作者。该研究得到了国家自然科学基金项目(32301897, 32070274, 31771882, 32072014, U20A2027, 31801389, 32472149)、黑龙江省重点研发计划项目(JD2023GJ01-07)和罗桂祥基金的资助。
论文链接:https://onlinelibrary.wiley.com/doi/full/10.1111/pce.15164
(供稿�/农学院)



